3.2 Práctica Guiada
3.2.1 Ejemplo 1: Iterando en la EPH
Lo primero que necesitamos es definir un vector o lista sobre el que iterar.
Por ejemplo, podemos armar un vector con los path a las bases individuales, con el comando fs::dir_ls
## fuentes/usu_individual_t119.txt fuentes/usu_individual_t418.txtLuego, como en la función que usamos para leer las bases definimos muchos parametros, nos podemos armar una función wrapper que sólo necesite un parámetro, y que simplifique la escritura del map
leer_base_eph <- function(path) {
read.table(path,sep=";", dec=",", header = TRUE, fill = TRUE) %>%
select(ANO4,TRIMESTRE,REGION,P21,CH04, CH06)
}
bases_df <- tibble(bases_individuales_path) %>%
mutate(base = map(bases_individuales_path, leer_base_eph))## # A tibble: 2 x 2
## bases_individuales_path base
## <fs::path> <list>
## 1 fuentes/usu_individual_t119.txt <df[,6] [59,369 × 6]>
## 2 fuentes/usu_individual_t418.txt <df[,6] [57,418 × 6]>El resultado es un DF donde la columna base tiene en cada fila, otro DF con la base de la EPH de ese período. Esto es lo que llamamos un nested DF o dataframe nesteado pa les pibes.
Si queremos juntar todo, podemos usar unnest()
## # A tibble: 116,787 x 7
## bases_individuales_path ANO4 TRIMESTRE REGION P21 CH04 CH06
## <fs::path> <int> <int> <int> <int> <int> <int>
## 1 fuentes/usu_individual_t119.txt 2019 1 41 0 2 28
## 2 fuentes/usu_individual_t119.txt 2019 1 41 0 2 13
## 3 fuentes/usu_individual_t119.txt 2019 1 41 0 1 1
## 4 fuentes/usu_individual_t119.txt 2019 1 41 5000 2 41
## 5 fuentes/usu_individual_t119.txt 2019 1 41 0 2 9
## 6 fuentes/usu_individual_t119.txt 2019 1 41 8000 1 51
## 7 fuentes/usu_individual_t119.txt 2019 1 41 0 1 63
## 8 fuentes/usu_individual_t119.txt 2019 1 41 0 2 62
## 9 fuentes/usu_individual_t119.txt 2019 1 41 0 2 24
## 10 fuentes/usu_individual_t119.txt 2019 1 41 3000 1 74
## # … with 116,777 more rows¿Qué pasa si los DF que tenemos nesteados no tienen la misma cantidad de columnas?
Esto mismo lo podemos usar para fragmentar el datastet por alguna variable, con el group_by()
## # A tibble: 6 x 2
## # Groups: REGION [6]
## REGION data
## <int> <list<df[,6]>>
## 1 41 [11,509 × 6]
## 2 44 [14,204 × 6]
## 3 42 [11,150 × 6]
## 4 43 [34,702 × 6]
## 5 40 [24,432 × 6]
## 6 1 [20,790 × 6]Así, para cada región tenemos un DF.
¿ De qué sirve todo esto?
No todo en la vida es un Dataframe. Hay estucturas de datos que no se pueden normalizar a filas y columnas. En esos casos recurríamos tradicionalmente a los loops. Con MAP podemos tener los elementos agrupados en un sólo objeto y aún conservar sus formas diferentes.
3.2.2 Ejemplo 2. Regresión lineal
Si bien no nos vamos a meter en el detalle del modelo lineal hoy, es útil usarlo como ejemplo de lo que podemos hacer con MAP.
Planteamos el modelo \[ P21 = \beta_0 + \beta_1*CH04 + \beta_2*CH06 \] Osea, un modleo que explica el ingreso según sexo y edad
##
## Call:
## lm(formula = P21 ~ factor(CH04) + CH06, data = bases_df)
##
## Residuals:
## Min 1Q Median 3Q Max
## -15472 -6606 -3367 2148 590198
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 4853.196 74.509 65.14 <2e-16 ***
## factor(CH04)2 -4063.112 72.200 -56.27 <2e-16 ***
## CH06 103.095 1.612 63.97 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 12300 on 116784 degrees of freedom
## Multiple R-squared: 0.05511, Adjusted R-squared: 0.0551
## F-statistic: 3406 on 2 and 116784 DF, p-value: < 2.2e-16(al final de la clase podemos charlar sobre los resultados, si hay interés :-) )
De forma Tidy, la librería broom nos da los resultados en un DF.
## # A tibble: 3 x 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) 4853. 74.5 65.1 0
## 2 factor(CH04)2 -4063. 72.2 -56.3 0
## 3 CH06 103. 1.61 64.0 0Si lo queremos hacer por region
3.2.2.1 Loopeando
resultados <- tibble()
for (region in unique(bases_df$REGION)) {
data <- bases_df %>%
filter(REGION==region)
lmfit <- lm(P21~factor(CH04)+CH06,data = data)
lmtidy <- broom::tidy(lmfit)
lmtidy$region <- region
resultados <- bind_rows(resultados,lmtidy)
}
resultados## # A tibble: 18 x 6
## term estimate std.error statistic p.value region
## <chr> <dbl> <dbl> <dbl> <dbl> <int>
## 1 (Intercept) 3768. 185. 20.3 3.15e- 90 41
## 2 factor(CH04)2 -3814. 180. -21.2 6.00e- 98 41
## 3 CH06 106. 4.18 25.3 1.12e-137 41
## 4 (Intercept) 7156. 291. 24.6 1.09e-130 44
## 5 factor(CH04)2 -5938. 278. -21.4 1.42e- 99 44
## 6 CH06 145. 6.32 23.0 1.40e-114 44
## 7 (Intercept) 4930. 231. 21.4 2.15e- 99 42
## 8 factor(CH04)2 -4007. 224. -17.9 1.71e- 70 42
## 9 CH06 97.8 4.95 19.7 2.68e- 85 42
## 10 (Intercept) 5107. 131. 39.0 0. 43
## 11 factor(CH04)2 -3949. 127. -31.1 5.02e-209 43
## 12 CH06 83.5 2.78 30.0 3.87e-195 43
## 13 (Intercept) 3329. 128. 26.0 4.12e-147 40
## 14 factor(CH04)2 -3239. 125. -25.9 3.74e-146 40
## 15 CH06 122. 2.89 42.2 0. 40
## 16 (Intercept) 5196. 197. 26.4 3.45e-151 1
## 17 factor(CH04)2 -4051. 189. -21.4 1.80e-100 1
## 18 CH06 88.2 4.12 21.4 1.98e-100 13.2.2.2 Usando MAP
Primero me armo una funcion que me simplifica el codigo
## # A tibble: 6 x 3
## # Groups: REGION [6]
## REGION data lm
## <int> <list<df[,6]>> <list>
## 1 41 [11,509 × 6] <tibble [3 × 5]>
## 2 44 [14,204 × 6] <tibble [3 × 5]>
## 3 42 [11,150 × 6] <tibble [3 × 5]>
## 4 43 [34,702 × 6] <tibble [3 × 5]>
## 5 40 [24,432 × 6] <tibble [3 × 5]>
## 6 1 [20,790 × 6] <tibble [3 × 5]>## # A tibble: 18 x 7
## # Groups: REGION [6]
## REGION data term estimate std.error statistic p.value
## <int> <list<df[,6]>> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 41 [11,509 × 6] (Intercept) 3768. 185. 20.3 3.15e- 90
## 2 41 [11,509 × 6] factor(CH0… -3814. 180. -21.2 6.00e- 98
## 3 41 [11,509 × 6] CH06 106. 4.18 25.3 1.12e-137
## 4 44 [14,204 × 6] (Intercept) 7156. 291. 24.6 1.09e-130
## 5 44 [14,204 × 6] factor(CH0… -5938. 278. -21.4 1.42e- 99
## 6 44 [14,204 × 6] CH06 145. 6.32 23.0 1.40e-114
## 7 42 [11,150 × 6] (Intercept) 4930. 231. 21.4 2.15e- 99
## 8 42 [11,150 × 6] factor(CH0… -4007. 224. -17.9 1.71e- 70
## 9 42 [11,150 × 6] CH06 97.8 4.95 19.7 2.68e- 85
## 10 43 [34,702 × 6] (Intercept) 5107. 131. 39.0 0.
## 11 43 [34,702 × 6] factor(CH0… -3949. 127. -31.1 5.02e-209
## 12 43 [34,702 × 6] CH06 83.5 2.78 30.0 3.87e-195
## 13 40 [24,432 × 6] (Intercept) 3329. 128. 26.0 4.12e-147
## 14 40 [24,432 × 6] factor(CH0… -3239. 125. -25.9 3.74e-146
## 15 40 [24,432 × 6] CH06 122. 2.89 42.2 0.
## 16 1 [20,790 × 6] (Intercept) 5196. 197. 26.4 3.45e-151
## 17 1 [20,790 × 6] factor(CH0… -4051. 189. -21.4 1.80e-100
## 18 1 [20,790 × 6] CH06 88.2 4.12 21.4 1.98e-100O incluso más facil, utilizando group_modify (que es un atajo que solo acepta DF)
## # A tibble: 18 x 6
## # Groups: REGION [6]
## REGION term estimate std.error statistic p.value
## <int> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 1 (Intercept) 5196. 197. 26.4 3.45e-151
## 2 1 factor(CH04)2 -4051. 189. -21.4 1.80e-100
## 3 1 CH06 88.2 4.12 21.4 1.98e-100
## 4 40 (Intercept) 3329. 128. 26.0 4.12e-147
## 5 40 factor(CH04)2 -3239. 125. -25.9 3.74e-146
## 6 40 CH06 122. 2.89 42.2 0.
## 7 41 (Intercept) 3768. 185. 20.3 3.15e- 90
## 8 41 factor(CH04)2 -3814. 180. -21.2 6.00e- 98
## 9 41 CH06 106. 4.18 25.3 1.12e-137
## 10 42 (Intercept) 4930. 231. 21.4 2.15e- 99
## 11 42 factor(CH04)2 -4007. 224. -17.9 1.71e- 70
## 12 42 CH06 97.8 4.95 19.7 2.68e- 85
## 13 43 (Intercept) 5107. 131. 39.0 0.
## 14 43 factor(CH04)2 -3949. 127. -31.1 5.02e-209
## 15 43 CH06 83.5 2.78 30.0 3.87e-195
## 16 44 (Intercept) 7156. 291. 24.6 1.09e-130
## 17 44 factor(CH04)2 -5938. 278. -21.4 1.42e- 99
## 18 44 CH06 145. 6.32 23.0 1.40e-114Pero MAP sirve para operar con cualquier objeto de R.
Por ejemplo podemos guardar el objeto S3:lm que es la regresion lineal entrenada. Ese objeto no es ni un vector, ni una lista, ni un DF. No es una estructura de datos, sino que es algo distinto, con propiedades como predict() para predecir, el summary() que vimos, etc.
fun<-function(porcion,grupo) { lm(P21~factor(CH04)+CH06,data = porcion)}
bases_df %>%
group_by(REGION) %>%
nest() %>%
mutate(lm = map(data,fun))## # A tibble: 6 x 3
## # Groups: REGION [6]
## REGION data lm
## <int> <list<df[,6]>> <list>
## 1 41 [11,509 × 6] <lm>
## 2 44 [14,204 × 6] <lm>
## 3 42 [11,150 × 6] <lm>
## 4 43 [34,702 × 6] <lm>
## 5 40 [24,432 × 6] <lm>
## 6 1 [20,790 × 6] <lm>3.2.3 Ejemplo 3: Gráficos en serie
Veamos un tercer ejemplo con otra base de datos que ya conocemos: Gapminder, que muestra algunos datos sobre la población de los países por año.
El objetivo de este ejercicio es hacer un gráfico por país de forma automática.
- Primero veamos los datos
## # A tibble: 10 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Italy Europe 2002 80.2 57926999 27968.
## 2 Sierra Leone Africa 2007 42.6 6144562 863.
## 3 United States Americas 1959 70.0 177830000 15240.
## 4 Lithuania FSU 2000 72.1 3654387 9632.
## 5 Bulgaria Europe 1993 71.2 8441872 6367.
## 6 Norway Europe 1953 73.2 3360888 10393.
## 7 Australia Oceania 2007 81.2 20434176 34435.
## 8 New Zealand Oceania 1950 69.3 1908310 11449.
## 9 Mongolia Asia 1972 53.8 1320500 1422.
## 10 France Europe 1953 67.3 42751746 7171.la base tiene la siguiente info:
- country: Nombre del país
- continent: Nombre del continente
- year: año
- lifeExp: Esperanza de vida al nacer
- pop: Población
gdpPercap
Vamos a hacer un gráfico sencillo para Argentina
data_argentina <- gapminder_unfiltered %>%
filter(country=='Argentina')
ggplot(data_argentina, aes(year, lifeExp, size= pop, color=gdpPercap))+
geom_point()+
geom_line(alpha=0.6)+
labs(title = unique(data_argentina$country))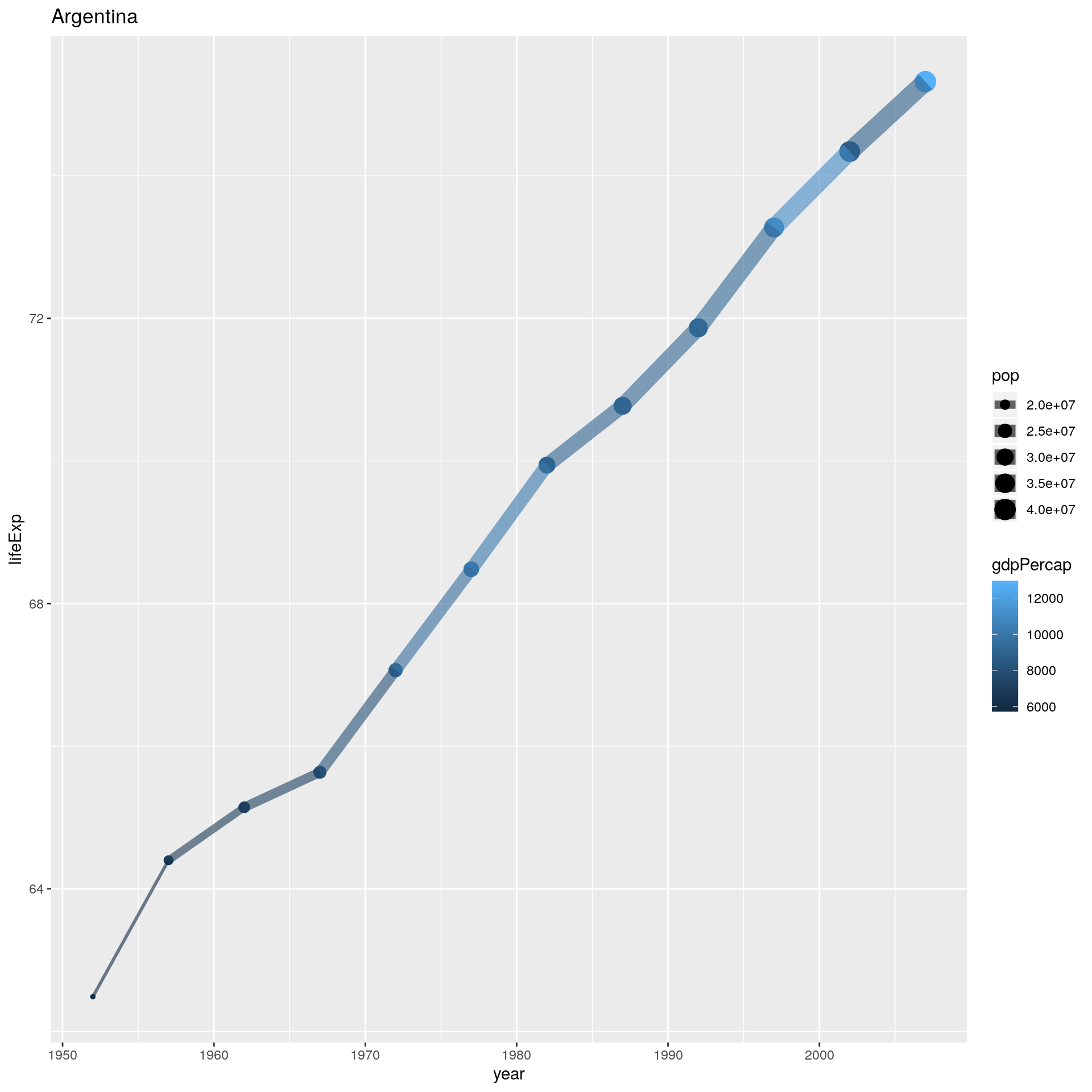
- Ahora que tenemos una idea de lo que queremos gráficar lo podemos poner adentro de una función que grafique.
# definimos la función
graficar_pais <- function(data, pais){
ggplot(data, aes(year, lifeExp, size= pop, color=gdpPercap))+
geom_point()+
geom_line(alpha=0.6)+
labs(title = pais)
}probamos la función para un caso
- Nos armamos un dataset nesteado
## # A tibble: 10 x 2
## # Groups: country [187]
## country data
## <fct> <list<df[,5]>>
## 1 Afghanistan [12 × 5]
## 2 Albania [12 × 5]
## 3 Algeria [12 × 5]
## 4 Angola [12 × 5]
## 5 Argentina [12 × 5]
## 6 Armenia [4 × 5]
## 7 Aruba [8 × 5]
## 8 Australia [56 × 5]
## 9 Austria [57 × 5]
## 10 Azerbaijan [4 × 5]- Ahora podemos crear una nueva columna que contenga los gráficos
gapminder_nest <- gapminder_nest %>%
mutate(grafico= map2(.x = data, .y = country,.f = graficar_pais))
gapminder_nest[1:10,]## # A tibble: 10 x 3
## # Groups: country [187]
## country data grafico
## <fct> <list<df[,5]>> <list>
## 1 Afghanistan [12 × 5] <gg>
## 2 Albania [12 × 5] <gg>
## 3 Algeria [12 × 5] <gg>
## 4 Angola [12 × 5] <gg>
## 5 Argentina [12 × 5] <gg>
## 6 Armenia [4 × 5] <gg>
## 7 Aruba [8 × 5] <gg>
## 8 Australia [56 × 5] <gg>
## 9 Austria [57 × 5] <gg>
## 10 Azerbaijan [4 × 5] <gg>Veamos un ejemplo
## [[1]]
Ahora podemos guardar todos los gráficos en un archivo PDF